linkage/fastlinkは、主に連鎖解析で使われるツール群で、ロッドスコアの計算に有用なプログラムが含まれています。 fastlinkはlinkageの一部のプログラムを最適化したものです。まずは試してみましょう。
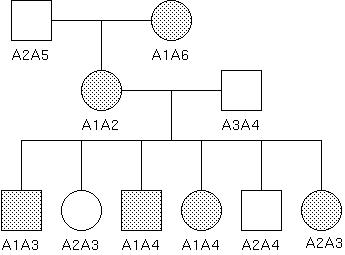
以下は『臨床ゲノム科学入門』(株式会社杏林図書)p.47図3-4 <ロッドスコア法による組み換え率の推定>からの引用です。

これをlinkageが解釈できる形式(Linkage format)にします。 Linkage formatは、例えばHaploviewなどのような他のプログラムでも 使える形式ですが、ここでは基本的な文法を説明します。Linkage formatは タブ区切りのテキストファイルで、一般的にタブで区切られたCSV形式として知られています。 そのため、作成には表計算ソフトを利用して作ることができます。
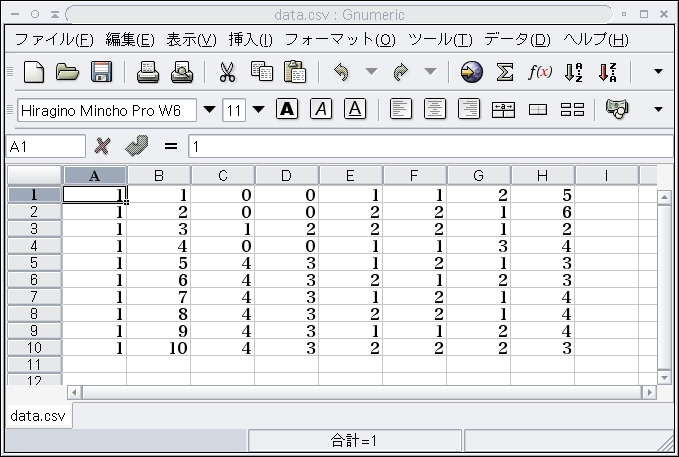
それぞれの行は個人をあらわし、A列は家系番号、B列は家系内の個人識別番号、 C列は父親の家系内個人式別番号(B列を参照する)、D列は母親の家系内個人識別番号 (同じくB列を参照する)、E列は性別(男=1、女=2)、F列は疾患 (unaffected=1、affected=2)、G列以降はアレルの組を次々に並べていきます。
作成できたら、makepedにCSVファイルを読ませて、pedfile.datを作成します。
MAKEPED Version 2.21
Usage: makeped pedigree.file output.file n
where the 'n' argument tells MAKEPED that
1) there are no loops
2) all probands should be chosen automatically
This version treats all ids as strings
but will use original pedigree ids if they ALL are integers
In addition, this version allows for multiple loops
Constants in effect
Maximum number of pedigrees500
Maximum number of individuals 4000
Maximum characters used in phenotypic data 200
Maximum number of characters in an id 11
Pedigree file -> data.csv
Output file -> pedfile.dat
Does your pedigree file contain any loops? (y/n) -> n
Do you want probands selected automatically? (y/n) -> y
これで以下のようなpedfile.datが生成されました。
1 1 0 0 3 0 0 1 1 1 2 5 Ped: 1 Per: 1
1 2 0 0 3 0 0 2 0 2 1 6 Ped: 1 Per: 2
1 3 1 2 5 0 0 2 0 2 1 2 Ped: 1 Per: 3
1 4 0 0 5 0 0 1 0 1 3 4 Ped: 1 Per: 4
1 5 4 3 0 6 6 1 0 2 1 3 Ped: 1 Per: 5
1 6 4 3 0 7 7 2 0 1 2 3 Ped: 1 Per: 6
1 7 4 3 0 8 8 1 0 2 1 4 Ped: 1 Per: 7
1 8 4 3 0 9 9 2 0 2 1 4 Ped: 1 Per: 8
1 9 4 3 0 10 10 1 0 1 2 4 Ped: 1 Per: 9
1 10 4 3 0 0 0 2 0 2 2 3 Ped: 1 Per: 10
また、pedfile.dat形式のファイルも自分で作ことができます。その場合、それぞれの 列の意味は以下の通りとなります。
次に解析内容を指示するdatafile.datを作成することになります。preplink.exeを利用して 作ることもできますが、非常に操作性が悪く、バグの温床になるので、ここでは エディタで作成します。まず今回のdatafile.datを以下に示します。
2 0 0 5 << NO. OF LOCI, RISK LOCUS, SEXLINKED (IF 1) PROGRAM@
0 0.0 0.0 0 << MUT LOCUS, MUT MALE, MUT FEM, HAP FREQ (IF 1)@
1 2
1 2 << AFFECTION, NO. OF ALLELES
0.99900 0.00100 << GENE FREQUENCIES
1 << NO. OF LIABILITY CLASSES
0 1.0000 1.0000 << PENETRANCES
3 6 << ALLELE NUMBERS, NO. OF ALLELES
0.25000 0.25000 0.2000 0.2000 0.05 0.05<< GENE FREQUENCIES
0 0 << SEX DIFFERENCE, INTERFERENCE (IF 1 OR 2)
0.00000000 << RECOMBINATION VALUES
1 0.05000 0.50000 << REC VARIED, INCREMENT, FINISHING VALUE
コメントは<<以降です。まず始めの行では、アレルの組の数+1、リスク、 性染色体連鎖、プログラム の種類(1=CILINK、2=CMAP、3=ILINK、4=LINKMAP、5=MLINK、6=LODSCORE、7=CLODSCORE) を指定します。次の行では変異の有無、男での変異率、女での変異率、連鎖平衡の有無を指定します。 3行目では、座の染色体上での順番を指定します。4行目以降は、それぞれのアレルまたは 疾患などについての記述となります。最後の3行に性別による違い、交差の干渉、 組み換えの頻度、ロッドスコアを どれだけ求めるかを指定します。ここでは、疾患とアレルの組についてのみ説明します。
抜粋
1 2 << AFFECTION, NO. OF ALLELES
0.99900 0.00100 << GENE FREQUENCIES
1 << NO. OF LIABILITY CLASSES
0 1.0000 1.0000 << PENETRANCES
疾患のあるなしの2つですので、最初の行は疾患解析を指定する1と2です。 次の行には遺伝子の頻度を指定しますが、家系内での大雑把な値で良いようです。 次に、疾患に関係するアレルの型の数を指定します。ここでは1つのみを指定しています。 次に、aa、aA、AAのそれぞれの遺伝子型での疾患の浸透率を指定します。 劣性であれば、0 0 1、優勢であれば0 1 1、それ以外にも指定できます。 もし遺伝子型が3つであれば、さきほどの行には3を、そして、浸透率は2行に分けて 11、12、13、22、23、33での浸透率をそれぞれ指定することになります。詳しくは ユーザガイドを参照して下さい。
抜粋
3 6 << ALLELE NUMBERS, NO. OF ALLELES
0.25000 0.25000 0.2000 0.2000 0.05 0.05<< GENE FREQUENCIES
アレルの指定番号である3とアレルの種類の数である6を最初の行に指定します。 次の行にそれぞれのアレルの頻度を指定します。
以上で、pedfile.dat、datafile.datが揃ったはずです。そうしたら、unkown.exeとmlink.exeを 続けて実行すると、outfile.datが生成されます。unkown.exe実行後には色々と文句をいわれるので 修正する必要がありますが、データファイルのエラーであることがあったり、浸透率や優性劣性の 指定を誤っていたりすることがあり、色々と苦労します。
LINKAGE (V5.1) WITH 2-POINT AUTOSOMAL DATA
ORDER OF LOCI: 1 2
-----------------------------------
-----------------------------------
THETAS 0.500
-----------------------------------
PEDIGREE | LN LIKE | LOG 10 LIKE
-----------------------------------
1 -30.678188 -13.323340 LOD= 0.000000
-----------------------------------
TOTALS -30.678188 -13.323340
-2 LN(LIKE) = 6.13564e+001 LOD SCORE = 0.000000
-----------------------------------
-----------------------------------
THETAS 0.000
-----------------------------------
PEDIGREE | LN LIKE | LOG 10 LIKE
-----------------------------------
1 -100000000000000000000.000000 -43429355638650388000.000000 LOD= -999.999999
-----------------------------------
TOTALS -100000000000000000000.000000 -43429355638650388000.000000
-2 LN(LIKE) = 2.00000e+020 LOD SCORE = -43429355638650388000.000000
-----------------------------------
-----------------------------------
THETAS 0.050
-----------------------------------
PEDIGREE | LN LIKE | LOG 10 LIKE
-----------------------------------
1 -29.771504 -12.929572 LOD= 0.393767
-----------------------------------
TOTALS -29.771504 -12.929572
-2 LN(LIKE) = 5.95430e+001 LOD SCORE = 0.393767
-----------------------------------
-----------------------------------
THETAS 0.100
-----------------------------------
PEDIGREE | LN LIKE | LOG 10 LIKE
-----------------------------------
1 -29.348693 -12.745948 LOD= 0.577391
-----------------------------------
TOTALS -29.348693 -12.745948
-2 LN(LIKE) = 5.86974e+001 LOD SCORE = 0.577391
-----------------------------------
-----------------------------------
THETAS 0.150
-----------------------------------
PEDIGREE | LN LIKE | LOG 10 LIKE
-----------------------------------
1 -29.229020 -12.693975 LOD= 0.629365
-----------------------------------
TOTALS -29.229020 -12.693975
-2 LN(LIKE) = 5.84580e+001 LOD SCORE = 0.629365
-----------------------------------
-----------------------------------
THETAS 0.200
-----------------------------------
PEDIGREE | LN LIKE | LOG 10 LIKE
-----------------------------------
1 -29.244461 -12.700681 LOD= 0.622659
-----------------------------------
TOTALS -29.244461 -12.700681
-2 LN(LIKE) = 5.84889e+001 LOD SCORE = 0.622659
-----------------------------------
-----------------------------------
THETAS 0.250
-----------------------------------
PEDIGREE | LN LIKE | LOG 10 LIKE
-----------------------------------
1 -29.344010 -12.743914 LOD= 0.579425
-----------------------------------
TOTALS -29.344010 -12.743914
-2 LN(LIKE) = 5.86880e+001 LOD SCORE = 0.579425
-----------------------------------
-----------------------------------
THETAS 0.300
-----------------------------------
PEDIGREE | LN LIKE | LOG 10 LIKE
-----------------------------------
1 -29.506653 -12.814549 LOD= 0.508790
-----------------------------------
TOTALS -29.506653 -12.814549
-2 LN(LIKE) = 5.90133e+001 LOD SCORE = 0.508790
-----------------------------------
-----------------------------------
THETAS 0.350
-----------------------------------
PEDIGREE | LN LIKE | LOG 10 LIKE
-----------------------------------
1 -29.723042 -12.908526 LOD= 0.414814
-----------------------------------
TOTALS -29.723042 -12.908526
-2 LN(LIKE) = 5.94461e+001 LOD SCORE = 0.414814
-----------------------------------
-----------------------------------
THETAS 0.400
-----------------------------------
PEDIGREE | LN LIKE | LOG 10 LIKE
-----------------------------------
1 -29.989724 -13.024344 LOD= 0.298996
-----------------------------------
TOTALS -29.989724 -13.024344
-2 LN(LIKE) = 5.99794e+001 LOD SCORE = 0.298996
-----------------------------------
-----------------------------------
THETAS 0.450
-----------------------------------
PEDIGREE | LN LIKE | LOG 10 LIKE
-----------------------------------
1 -30.306998 -13.162134 LOD= 0.161206
-----------------------------------
TOTALS -30.306998 -13.162134
-2 LN(LIKE) = 6.06140e+001 LOD SCORE = 0.161206
-----------------------------------
-----------------------------------
THETAS 0.500
-----------------------------------
PEDIGREE | LN LIKE | LOG 10 LIKE
-----------------------------------
1 -30.678188 -13.323340 LOD= 0.000000
-----------------------------------
TOTALS -30.678188 -13.323340
-2 LN(LIKE) = 6.13564e+001 LOD SCORE = 0.000000
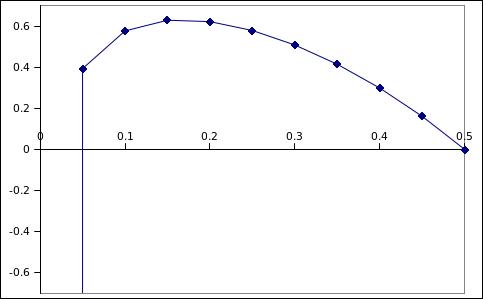
ロッドスコアが求められていることが分かります。試しにグラフをかくと 以下のようになりますので、おそらく0.15あたりが妥当だと考えられます。
fastlinkには、unkown.exeやmlink.exe等最小限のプログラムしか入っていないので、 makepedやpreplinkを実行するにはlinkageのsupplement programが必要になります。 fastlinkは NCBI FASTLINK Home Pageから、linkageは FTP directory of linkageから 手に入れられますし、その他のsoftware群については、 linkage software list で調べることができます。ただし、どれも研究で作られたもののため あまりメンテナンスされていないのが現状です。
<rindolf> Hi all.
<rindolf> LeoNerd: here?
<LeoNerd> rindolf: Maybe
<rindolf> LeoNerd: "be here or be not here - there is no maybe"
<LeoNerd> :)
<rindolf> LeoNerd: a.k.a the law of the exclusion of the middle.
<dwu> I think Yoda phrased that one best.
-- To be here or not to be here on Freenode's #perl
-- #perl, Freenode
I fell asleep reading a dull book, and I dreamt that I was reading on,
so I woke up from sheer boredom.